Praktische Anwendungen für die Landwirtschaft
Im zweiten Teil dieser Serie hat Robert die Geschichte der Pflanzenzüchtung zusammenfassend dargestellt und aufgezeigt, welche Werkzeuge im Laufe der Zeit entwickelt wurden, um Pflanzen züchterisch zu verbessern. Auch in Zukunft wird die Pflanzenzüchtung eine tragende Rolle spielen, wenn es darum geht, die Herausforderungen in der Landwirtschaft zu meistern. Ich denke in diesem Punkt besteht Einigkeit zwischen allen Interessengruppen der landwirtschaftlichen Wertschöpfungskette. Über die Frage, welche Züchtungsmethoden eingesetzt werden sollen, um sich diesen Herausforderungen zu stellen, gehen die Meinungen hingegen weit auseinander… Im dritten und letzten Teil dieser Serie geht es um ein in der jüngeren Vergangenheit entwickeltes Werkzeug, das großes Potential hat, verbesserte Sorten zu entwickeln, aber trotzdem sehr kontrovers diskutiert wird: Das Genome Editing.Genome Editing ist ein Sammelbegriff für eine Reihe von Züchtungstechniken1, mit denen das Genom unter anderem von Pflanzen an einer ganz spezifischen Stelle verändert werden kann. Auf die technischen Feinheiten möchte ich jedoch nicht eingehen, sondern hierfür auf bereits existierende Artikel verweisen23. In diesem Beitrag möchte ich in einem ersten Schritt einen Überblick über den Stand der Entwicklung von Genome Editing geben, globale Trends aufzeigen und darstellen, welche marktorientierten Anwendungen bereits existieren. Im Anschluss werde ich an vier konkreten Anwendungsbeispielen aufzeigen, was mit Genome Editing bereits jetzt möglich ist und welche Vorteile diese Art der Züchtung gegenüber etablierten Techniken hat. Grundlage für diesen Beitrag ist ein Teil meiner Promotion, bei der ich zusammen mit meinen Arbeitskollegen eine systematische Literaturrecherche zu Genome Editing durchgeführt habe 45. Die Literatursuche fand im Mai 2018 statt, weshalb spätere Veröffentlichungen hier nicht berücksichtigt werden.
CRISPR/Cas auf dem Vormarsch
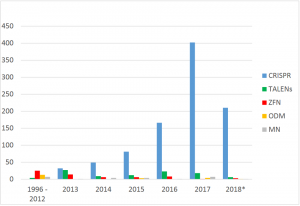
Im Zeitraum zwischen 1996 und Mai 2018 wurden über 1300 Anwendungen identifiziert, in denen eine der Genome Editing Techniken eingesetzt wurde, um eine Veränderung im Genom einer Modell-, Kultur- oder Zierpflanze zu erzeugen.
Wie aus Abbildung 1 erkennbar ist, wurden im Zeitraum zwischen 1996 und 2012 nur wenige Anwendungen zu Genome Editing veröffentlicht. Seit der Entwicklung des CRISPR/Cas-Systems und dessen erster Veröffentlichung im Jahr 2013 in Pflanzen, ist die Anzahl der Anwendungen exponentiell angestiegen. Allein im Jahr 2017 konnten über 400 Anwendungen identifiziert werden, die mit der CRISPR/Cas Technik durchgeführt wurden. Dies liegt hauptsächlich daran, dass die Technik im Vergleich zu den anderen Genome Editing Techniken deutlich schneller und kostengünstiger eingesetzt werden kann.
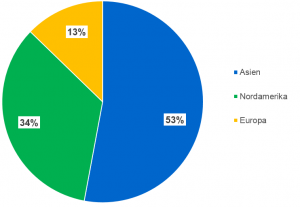
Europa hinkt bei der Entwicklung hinterher
Betrachtet man die globale Verteilung der Anwendungen fällt auf, dass 53% in Asien (hauptsächlich China) und 34% in Nordamerika (hauptsächlich USA) veröffentlicht wurden. Lediglich 13% der Anwendungen wurden in Europa durchgeführt (die Hälfte davon in Deutschland; Abbildung 2). Dies zeigt deutlich, dass Europa im weltweiten Vergleich bereits heute weit hinter den führenden Nationen liegt. Da die Datenerhebung vor dem Urteil des Europäischen Gerichtshofs stattgefunden hat, sind die Auswirkungen der strengen europäischen Regulierung und der weniger strengen Regulierungen in weiten Teilen der Welt (wie zum Beispiel in den USA oder Kanada) hier noch nicht wiedergespiegelt. Insgesamt konnten Anwendungen aus 33 Ländern identifiziert werden, von denen nur sehr wenige aus Afrika, Südamerika und Australien kommen (jeweils unter 1% der Anwendungen).
Hauptsächlich Punktmutationen
Des Weiteren hat die Untersuchung ergeben, dass in über 1200 Anwendungen (95%) gezielt Punktmutationen im Genom der Pflanzen erzeugt wurden. Diese Punktmutationen sind vom Ergebnis (Endprodukt) her vergleichbar mit natürlich auftretenden Mutationen (siehe hierzu den Artikel von Anna) und künstlich induzierten Mutationen durch Strahlung oder Chemikalien (siehe hierzu den Artikel von Robert). Nur bei den wenigen restlichen Anwendungen wurden arteigene (Cisgene) oder artfremde (Transgene) Gene eingefügt. Diese Anwendungen sind vergleichbar mit der klassischen Gentechnik, mit dem Unterschied, dass das Cis- oder Transgen beim Genome Editing nicht an einer zufälligen, sondern einer gezielten Stelle im Genom eingefügt wird.
Pflanzen mit neuen Eigenschaften
Der größte Teil der bisher identifizierten Anwendungen kann als Grundlagenforschung eingestuft werden. Das bedeutet, dass Genome Editing in einer Modellpflanze und nicht in einer Nutzpflanze angewendet wurde oder das Gene adressiert wurden, die keine praktische Bedeutung für die Landwirtschaft haben. Häufig wurden auch sog. „Genfunktionsanalysen“ durchgeführt, mit deren Hilfe man die Funktion von Genen besser verstehen will. Nichtsdestotrotz identifizierten wir bis Mai 2018 knapp 100 marktorientierte Anwendungen. Diese können verschiedenen Kategorien zugeordnet werden (Abbildung 3).
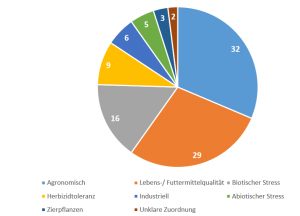
1. Verbesserte Lebens- und Futtermittelqualität:
2. Agronomische Eigenschaften
3. Resistenz gegen abiotischen Stress
4. Resistenzen gegen biotischen Stress
- Toleranz gegen die Pilzerkrankung „Phytophtora tropicalis“ in Kakao12
- Mehltautoleranz in Tomate13 und Weizen14
5. Herbizidtoleranz
6. Industrielle Nutzung
- Reduzierter Ligningehalt in Zuckerrohr (Zur Biokraftstoffproduktion)17
- Veränderte Stärkezusammensetzung in Kartoffel18
Eine detaillierte Auflistung aller marktorientierter Anwendungen kann hier eingesehen werden.
Die nach unserem Wissen erste Sorte, die kommerziell angebaut wird, ist eine Sojabohne mit veränderter Ölzusammensetzung, die einen höheren Anteil an gesunden Fetten aufweist. Die entsprechende Sorte wurde von der Firma Calyxt entwickelt und wird in den USA seit Anfang 2019 vermarktet.
Das breite Spektrum der Anwendungen, die bereits weite Teile der landwirtschaftlichen Wertschöpfungskette abdecken und in 28 verschiedenen Kulturarten identifiziert wurden, zeigt, dass Genome Editing ein vielversprechendes Werkzeug ist, um neue Sorten zu züchten. Diese können besser an die Bedürfnisse der Landwirtschaft angepasst sein und dadurch einen wertvollen Beitrag zur Ernährungssicherheit und einer nachhaltigen Landwirtschaft leisten.
Anwendungsbeispiele
Im Folgenden möchte ich nun an vier konkreten Anwendungsbeispielen aufzeigen, was mit Genome Editing bereits jetzt möglich ist und welche Vorteile diese Art der Züchtung gegenüber etablierten Züchtungstechniken hat.
Anwendung 1: Reduzierter Arsengehalt in Reis

Arsen ist ein chemisches Element, welches natürlicherweise in der Erdkruste vorhanden ist und über das Grundwasser in unsere Nahrung gelangt. Da Reis meist auf überfluteten Feldern angebaut wird, ist er besonders anfällig für eine Arsenanreicherung in der Pflanze und den Körnern. Arsenrückstände verursachen bei übermäßiger Einnahme gesundheitliche Probleme wie Hautveränderungen, Gefäß- und Nervenschädigungen, bis hin zu Krebserkrankungen. Dies ist der Grund dafür, dass es für anorganisches Arsen in der EU-Grenzwerte von 0,2 bis 0,3 mg/kg gibt. In Lebensmitteln für Säuglinge dürfen sogar nur 0,1 mg/kg anorganisches Arsen enthalten sein. Neben veränderten Anbaubedingungen, die jedoch mit einem Ertragsverlust einhergehen, spielt die Sortenwahl eine entscheidende Rolle bei der Arsenreduzierung. Deshalb rät die WHO Reissorten zu züchten, die weniger Arsen aufnehmen19. Chinesische Wissenschaftler der Sun Yat-sen Universität haben ein Gen identifiziert, welches für den Arsentransport in die Pflanze verantwortlich ist. Dieses Gen wurde mit der CRISPR/Cas Technik ausgeschaltet, wodurch der Transport von Arsen in die Pflanze und die Einlagerung im Reiskorn verringert wurden 2021. Die Ergebnisse liefern wichtige Erkenntnisse, um Sorten mit verminderten Arsenrückständen zu züchten.
Es gibt bereits heute konventionell gezüchtete Sorten, die eine erhöhte Arsentoleranz aufweisen. Hat man jedoch eine Reissorte A mit guten Ertrags- und Qualitätseigenschaften, die aber keine Arsentoleranz besitzt, hat man zwei Möglichkeiten: 1. Man benutzt Genome Editing in Reissorte A, um – wie oben beschrieben – direkt die Arsentoleranz in der Pflanze zu bekommen oder 2. Man kreuzt die Reissorte A mit einer arsentoleranten Sorte B. Das Problem bei letzterem Vorgehen ist jedoch, dass neben der gewünschten Eigenschaft (nach den Regeln der Vererbungslehre) die Tochtergeneration (also das Produkt der Kreuzung) jeweils die Hälfte der Gene von Sorte A und Sorte B in sich trägt. Um alle ungewünschten Eigenschaften der Wildsorte wieder loszuwerden, kann man Pflanzen „rückkreuzen“. Das bedeutet, dass man die Tochtergeneration wieder mit der Ausgangssorte A kreuzt. Diesen Schritt der Rückkreuzung führt man mehrere Male, jeweils mit der Ausgangssorte A, durch und selektiert stets auf die Eigenschaft der Arsentoleranz. Nach circa sieben Generationen erhält man eine Reissorte, die (fast) alle Eigenschaften der Reissorte A besitzt und zusätzlich die Eigenschaft der Arsentoleranz. Wie anhand dieser Ausführung gut zu erkennen ist, benötigt diese Methode viele Jahre und ist mit viel Arbeits- und Selektionsaufwand (zur Identifizierung der arsentoleranten Pflanzen) verbunden. Mit Genome Editing kann man das gleiche Ergebnis in viel kürzerer Zeit erreichen, weil man das gewünschte Gen eben direkt einbaut und den Rest des Genoms unbehelligt lässt. Das Endprodukt unterscheidet sich unabhängig von der gewählten Methode nicht voneinander.
Anwendung 2: Veränderte Stärkezusammensetzung in Kartoffel

Neben dem Anbau von Kartoffeln als Nahrungsmittel, wird Kartoffelstärke auch industriell verwendet. Stärke (zählt zu den Kohlenhydraten) ist ein wesentlicher Bestandteil in Kartoffeln und besteht aus zwei Komponenten: Amylopektin (zu ca.75%) und Amylose (zu ca. 25%). Für die industrielle Stärkenutzung, z.B. für Kleb- und Schmierstoffe, wird lediglich Amylopektin benötigt. Deshalb müssen die beiden Stärketypen voneinander getrennt werden. Dies ist mit chemischen, physikalischen oder enzymatischen Verfahren zwar möglich, jedoch arbeitsintensiv und kann je nach Verfahren zu Umweltbelastungen führen. Wissenschaftlern der Swedish University of Agricultural Sciences ist es gelungen, mithilfe von CRISPR/Cas ein Gen auszuschalten, das für die Amylosebildung verantwortlich ist. Das Resultat ist eine Kartoffel, die ausschließlich Amylopektin bildet, wodurch der anschließende Trennungsaufwand entfällt 22.
Hier ist anzumerken, dass es bereits die konventionell gezüchtete Kartoffelsorte Eliane gibt, die durch ungerichtete Mutagenese (Strahlung) erzeugt wurde. Bei der ungerichteten Mutagenese wird Saatgut mit Chemikalien oder Strahlung behandelt, was unspezifische Mutationen an vielen Stellen im Genom auslöst (siehe Tabelle 1). Um daraus wieder vitale Pflanzen zu bekommen, sind auch hier aufwendige Rückkreuzungsarbeiten notwendig. Das Endprodukt ist unabhängig von der verwendeten Züchtungstechnik eine Kartoffel, die ausschließlich Amylopektin bildet.
Die folgende Tabelle stellt die wesentlichen Unterschiede zwischen Genome Editing und der ungerichteten Mutagenese dar:
Tabelle 1: Unterschiede zwischen Genome Editing und der ungerichteten Mutagenese
Genome Editing | Ungerichtete Mutagenese | |
| Anzahl Mutationen | Eine | Sehr viele |
| Spezifität der Mutation(en) | An einer spezifischer Stelle im Genom | An (vielen) unspezifischen Stellen im Genom |
| Anzahl benötigter Pflanzen | Wenige Pflanzen werden benötig (aufgrund hoher Mutationsrate des Zielgens) | Sehr viele Pflanzen werden benötigt (aufgrund sehr geringer Mutationsrate des Zielgens und schlechter Vitalität der Pflanze) |
| Ungewollte Mutationen | (meist) keine ungewollten Mutationen | Viele ungewollte Mutationen an anderen Stellen im Genom |
| Rückkreuzungen nötig, um vitale Pflanzen zu generieren | Nein | Ja |
| Benötigte Zeit | Monate | Viele Jahre |
Quelle: Eigene Darstellung.
Wie der Europäische Gerichtshof im vergangenen Jahr geurteilt hat, müssen alle Genom-editierten Pflanzen nach den Gentechnikregelungen zugelassen werden, was sehr zeitaufwendig und mit enormen Kosten verbunden ist (was das genau bedeutet, werde ich in einem späteren Artikel genauer beleuchten). Werden Sorten dagegen mithilfe der ungerichteten Mutagenese gezüchtet, sind keine besonderen Sicherheitsprüfungen notwendig und die entsprechenden Sorten müssen lediglich den normalen Sortenzulassungsprozess durchlaufen.
Anwendung 3: Toleranz gegen die Grauschimmelfäule in Wein

Grauschimmel („Botrytis cinerea“) ist ein weit verbreiteter Schimmelpilz, der als Schädling über 2035 Wirtspflanzen befällt. Im Weinbau kann es bei feuchter Witterung oder bei Verletzung der Beeren beispielsweise infolge von Hagel, starken Niederschlägen oder Sonnenbrand (ja, auch Pflanzen können einen Sonnenbrand bekommen!) zu Problemen mit Botrytis cinerea kommen. Befällt der Pilz die unreifen Beeren, verursacht er Ertrags- und Qualitätseinbußen. Vor allem weichschalige Sorten wie Müller-Thurgau und Riesling sind anfällig. Kommt es trotz vorbeugenden Kulturmaßnahmen wie ausreichender Belüftung zum Befall, können als letztes Mittel Fungizide wie Teldor im konventionellen Anbau (bis zu zwei Anwendungen im Jahr) oder Kaliumhydrogencarbonat im Ökolandbau (bis zu sechs Anwendungen im Jahr) eingesetzt werden2324.
Wissenschaftlern der Northwest A&F University in China ist es gelungen, mithilfe der CRISPR/Cas Technik gezielt eine Punktmutation in einem Gen zu erzeugen, das für seine tragende Rolle bei der biotischen Stressabwehr bekannt ist. Die Ergebnisse des Versuchs lieferten eine erhöhte Toleranz gegen den Grauschimmel25. Durch solche Ansätze kann der Einsatz von Pflanzenschutzmitteln reduziert werden und ein Beitrag für eine umweltschonendere Landwirtschaft geleistet werden.
Wie bereits im ersten Beispiel erwähnt, hat man auch hier die Möglichkeit das Resistenzgen aus einer Wildart einzukreuzen. Am Beispiel Reis dauern die benötigten sieben Rückkreuzungen sieben Jahre. Bei Wein, der erst im dritten Jahr blüht und erst dann rückgekreuzt werden kann, dauern sieben Rückkreuzung schon 21 Jahre. Erschwerend kommt hierbei hinzu, dass selbst nach sieben Rückkreuzungen noch „Reste“ der Wildart in der neu gezüchteten Sorte verbleiben, die den Geschmack der neu gezüchteten Sorte verfälschen können. Ein Unding für jeden Weingourmet…
Anwendung 4: Resistenz gegen CBSD Virus in Maniok
Maniok ist ein wichtiges Grundnahrungsmittel in afrikanischen Ländern wie Burundi, Ruanda, Uganda, Kenia und Tansania und wird dort von vielen Kleinbauern angebaut. Es ist nach Mais, Reis, Weizen und Kartoffel die weltweit am häufigsten angebaute Nutzpflanze. Im Jahr 2011 hat die UN Food and Agriculture Organization (FAO) vor der Ausbreitung eines Pflanzenvirus gewarnt, das große Teile der Ernte und damit die Lebensgrundlage der Bevölkerung vernichtet. Das Virus ist verantwortlich für die Braunstreifenkrankheit (Cassava Brown Streak Disease (CBSD))26. Es verfärbt die Maniokknolle bräunlich, wodurch sie ungenießbar wird. Das tückische daran ist, dass man den Pflanzenteilen, die über der Erde liegen, die Virusinfektion nicht ansieht und der Schaden erst nach der Ernte sichtbar wird.
In einem von der Bill & Melinda Gates Foundation unterstützen Arbeit, schafften es Forscher der University of California, durch das Ausschalten mehrerer Gene durch CRISPR/Cas, Maniokpflanzen zu erzeugen, die eine erhöhte Toleranz gegen die Braunstreifenkrankheit aufweisen27.
Da das Thema Gentechnik in Entwicklungsländern sehr komplex ist, möchte ich an dieser Stelle Herr Professor Martin Qaim, Professor für Internationale Ernährungswirtschaft und Ländliche Entwicklung, von der Uni Göttingen zitieren, der die wesentlichen Aspekte sehr gut zusammengefasst hat:
„Europäer können es sich leisten, gentechnisch veränderte Lebensmittel abzulehnen, Menschen auf anderen Kontinenten nicht. (…) Es geht gar nicht darum, dass die Gentechnik letztlich irgendwie der Schlüssel zur Lösung aller Probleme wäre, aber Gentechnik oder Genome Editing (…) bietet schon große Potenziale, tatsächlich Pflanzen hervorzubringen, die widerstandsfähiger sind, auch gegen zunehmenden Klimastress. (…) Was wir in Afrika jedoch sicherlich nicht brauchen, sind ganz, ganz starke Patente, also da warne ich vor. (…) Wir werden nicht umhinkommen, öffentliche Agrarforschung zu fördern, damit das, was im Kleinbauernsektor benötigt wird, auch tatsächlich entwickelt wird und dann auch von den Preisen her zugänglich für diese Bauern ist.“28
Abschließend möchte ich noch meine persönliche Meinung darstellen:
Für eine progressive Agrarwende ist es essentiell hohe und stabile Erträge zu sichern und gleichzeitig die negativen Auswirkungen der Landwirtschaft auf die Umwelt und Artenvielfalt soweit wie möglich zu reduzieren. Dazu sollte man für jedes Problem überlegen, welche Methoden am besten geeignet sind, um diese zu lösen – anstatt einzelne Techniken von vornherein abzulehnen und lediglich mit den verbleibenden die Probleme anzugehen.
(c) Titelbild: Henrike Perner
- Die CRISPR-Bibliothek ist da! - 5. April 2020
- CRISPR Advent Calendar - 29. November 2019
- CRISPR-Adventskalender - 28. November 2019
Einzelnachweise
- Genome Editing ist der Überbegriff für folgende Züchtungstechniken: CRISPR/Cas, TALENs, Zink-Finger Nukleasen (ZFN), Meganukleasen (MN) und Oligonukleotid-gerichtete Mutagenese (ODM)
- Müllner A. CRISPR/Cas – Die Grundlagen. 2019. https://progressive-agrarwende.org/crispr-cas-grundlagen/. Accessed 11 Jun 2019.
- Modrzejewski D, Sprink T, Hartung F. DIALOG GEA | 2017-07-10 Mega, Crispr, Finger? Die Techniken des Genome Editings. https://www.dialog-gea.de/de/themen/inhalte/2017-07-10-mega-crispr-finger-die-techniken-des-genome-editings. Accessed 11 Jun 2019.
- Modrzejewski D, Hartung F, Sprink T, Krause D, Kohl C, Schiemann J, Wilhelm R. What is the available evidence for the application of genome editing as a new tool for plant trait modification and the potential occurrence of associated off-target effects: a systematic map protocol. Environ Evid. 2018;7:11. doi:10.1186/s13750-018-0130-6
- Modrzejewski D, Hartung F, Sprink T, Krause D, Kohl C, Wilhelm R. 1. Aktualisierung der Übersicht über Nutz‐ und Zierpflanzen, die mittels neuer molekularbiologischer Techniken für die Bereiche Ernährung, Landwirtschaft und Gartenbau erzeugt wurden. https://www.bmel.de/SharedDocs/Downloads/Landwirtschaft/Pflanze/GrueneGentechnik/NMT_Stand-Regulierung_Anlage4-Aktualisierung.pdf;jsessionid=16E20F7BC1822B0694CA6D0EC1C5864A.1_cid376?__blob=publicationFile. Accessed 20 Jun 2019.
- Haun W, Coffman A, Clasen BM, Demorest ZL, Lowy A, Ray E, et al. Improved soybean oil quality by targeted mutagenesis of the fatty acid desaturase 2 gene family. Plant Biotechnol J. 2014;12:934–40. doi:10.1111/pbi.12201.
- Sánchez-León S, Gil-Humanes J, Ozuna CV, Giménez MJ, Sousa C, Voytas DF, Barro F. Low-gluten, nontransgenic wheat engineered with CRISPR/Cas9. Plant Biotechnol J 2017. doi:10.1111/pbi.12837.
- Wang W, Pan Q, He F, Akhunova A, Chao S, Trick H, Akhunov E. Transgenerational CRISPR-Cas9 Activity Facilitates Multiplex Gene Editing in Allopolyploid Wheat. The CRISPR Journal. 2018;1:65–74. doi:10.1089/crispr.2017.0010.
- Hu B, Li D, Liu X, Qi J, Gao D, Zhao S, et al. Engineering Non-transgenic Gynoecious Cucumber Using an Improved Transformation Protocol and Optimized CRISPR/Cas9 System. Mol Plant. 2017;10:1575–8. doi:10.1016/j.molp.2017.09.005.
- Shi J, Gao H, Wang H, Lafitte HR, Archibald RL, Yang M, et al. ARGOS8 variants generated by CRISPR-Cas9 improve maize grain yield under field drought stress conditions. Plant Biotechnol J. 2017;15:207–16. doi:10.1111/pbi.12603.
- Duan Y-B, Li J, Qin R-Y, Xu R-F, Li H, Yang Y-C, et al. Identification of a regulatory element responsible for salt induction of rice OsRAV2 through ex situ and in situ promoter analysis. Plant Mol Biol. 2016;90:49–62. doi:10.1007/s11103-015-0393-z.
- Fister AS, Landherr L, Maximova SN, Guiltinan MJ. Transient Expression of CRISPR/Cas9 Machinery Targeting TcNPR3 Enhances Defense Response in Theobroma cacao. Front. Plant Sci. 2018;9:47. doi:10.3389/fpls.2018.00268.
- Nekrasov V, Wang C, Win J, Lanz C, Weigel D, Kamoun S. Rapid generation of a transgene-free powdery mildew resistant tomato by genome deletion. Sci Rep. 2017;7:482. doi:10.1038/s41598-017-00578-x.
- Wang Y, Cheng X, Shan Q, Zhang Y, Liu J, Gao C, Qiu J-L. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew. Nat Biotechnol. 2014;32:947–51. doi:10.1038/nbt.2969.
- Sauer NJ, Narváez-Vásquez J, Mozoruk J, Miller RB, Warburg ZJ, Woodward MJ, et al. Oligonucleotide-Mediated Genome Editing Provides Precision and Function to Engineered Nucleases and Antibiotics in Plants. Plant Physiol. 2016;170:1917–28. doi:10.1104/pp.15.01696.
- D’Halluin K, Vanderstraeten C, van Hulle J, Rosolowska J, van den Brande I, Pennewaert A, et al. Targeted molecular trait stacking in cotton through targeted double-strand break induction. Plant Biotechnol J. 2013;11:933–41. doi:10.1111/pbi.12085.
- Jung JH, Altpeter F. TALEN mediated targeted mutagenesis of the caffeic acid O-methyltransferase in highly polyploid sugarcane improves cell wall composition for production of bioethanol. Plant Mol Biol. 2016;92:131–42. doi:10.1007/s11103-016-0499-y.
- Andersson M, Turesson H, Nicolia A, Fält A-S, Samuelsson M, Hofvander P. Efficient targeted multiallelic mutagenesis in tetraploid potato (Solanum tuberosum) by transient CRISPR-Cas9 expression in protoplasts. Plant Cell Rep. 2017;36:117–28. doi:10.1007/s00299-016-2062-3.
- UGB, Gesundheitsberatung Vfu. Belastung mit Arsen – ein ungelöstes Problem – UGB-Gesundheitsberatung. 04.10.2017. https://www.ugb.de/lebensmittel-im-test/belastung-mit-arsen/. Accessed 20 Jun 2019.
- Wang F-Z, Chen M-X, Yu L-J, Xie L-J, Yuan L-B, Qi H, et al. OsARM1, an R2R3 MYB Transcription Factor, Is Involved in Regulation of the Response to Arsenic Stress in Rice. Front Plant Sci. 2017;8:1868. doi:10.3389/fpls.2017.01868.
- Ye Y, Li P, Xu T, Zeng L, Cheng D, Yang M, et al. OsPT4 Contributes to Arsenate Uptake and Transport in Rice. Front. Plant Sci. 2017;8:311. doi:10.3389/fpls.2017.02197.
- Andersson M, Turesson H, Nicolia A, Fält A-S, Samuelsson M, Hofvander P. Efficient targeted multiallelic mutagenesis in tetraploid potato (Solanum tuberosum) by transient CRISPR-Cas9 expression in protoplasts. Plant Cell Rep. 2017;36:117–28. doi:10.1007/s00299-016-2062-3.
- Grauschimmel, Botrytis (Botrytis cinerea) an Hausrebstock und Tafeltraube. https://www.lwg.bayern.de/gartenakademie/gartendokumente/infoschriften/079138/index.php. Accessed 20 Jun 2019.
- Grauschimmel (Botrytis cinerea) Sauerfäule Rohfäule Bodentrauben Edelfäule. 10.12.2018. https://www.oekolandbau.de/landwirtschaft/pflanze/grundlagen-pflanzenbau/pflanzenschutz/schaderreger/schadorganismen-im-weinbau/pilzkrankheiten/grauschimmel-botrytis-cinerea-sauerfaeule-rohfaeule-bodentrauben-edelfaeule/. Accessed 20 Jun 2019.
- Wang X, Tu M, Wang D, Liu J, Li Y, Li Z, et al. CRISPR/Cas9-mediated efficient targeted mutagenesis in grape in the first generation. Plant Biotechnol J. 2018;16:844–55. doi:10.1111/pbi.12832.
- FAO – News Article: Cassava virus on verge of epidemic in East Africa. http://www.fao.org/news/story/en/item/94313/icode/. Accessed 19 Jun 2019.
- Gomez MA, Lin ZD, Moll T, Chauhan RD, Hayden L, Renninger K, et al. Simultaneous CRISPR/Cas9-mediated editing of cassava eIF4E isoforms nCBP-1 and nCBP-2 reduces cassava brown streak disease symptom severity and incidence. Plant Biotechnol J. 2019;17:421–34. doi:10.1111/pbi.12987
- Landwirtschaft in Afrika – „Gentechnik bietet große Potenziale“. 29.11.2017. https://www.deutschlandfunkkultur.de/landwirtschaft-in-afrika-gentechnik-bietet-grosse-potenziale.1008.de.html?dram:article_id=401861. Accessed 23 Jun 2019.

3 Kommentare